pyimageJ教程(2) 初识PyImageJ
- 获取链接
- X
- 电子邮件
- 其他应用
pyimageJ教程(2) 初识PyImageJ
PyImageJ 与传统 ImageJ 的技术对比
「功能扩展性:」 传统 ImageJ 拥有庞大的插件生态(如Fiji自带数百种插件)和多种脚本语言支持(宏语言、Beanshell、JavaScript、Groovy、Jython 等),能满足常见生物图像处理需求。PyImageJ 则将 ImageJ/Fiji 的所有功能暴露给 Python 环境,用户可以直接调用 ImageJ2 的完整 API 及其插件,也可以利用 ImageJ1 的兼容层继续使用旧插件。这种桥接允许在同一程序中混用 ImageJ 的各种算法与 Python 生态中的工具(如 NumPy、SciPy、scikit-image、OpenCV、ITK 等)。例如,PyImageJ 可以调用 ImageJ 的 Find Maxima 等算法进行图像分析(下图示例演示了利用 PyImageJ 在细胞显微图像上执行 Find Maxima 的结果),同时也能结合 Python 库扩展功能。
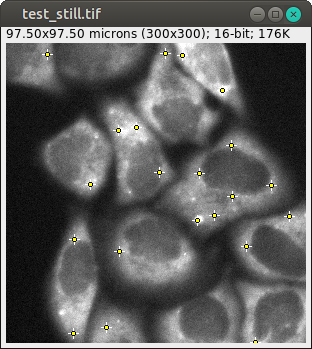
图:PyImageJ 调用 ImageJ 的 Find Maxima 功能对细胞图像进行中心点检测。PyImageJ 将 ImageJ2 的所有功能(包括细胞检测算法)桥接到 Python 中。
「可编程性:」 传统 ImageJ 通过脚本编辑器支持多种内置脚本语言,如易学但功能受限的宏语言,或基于 Java 的 Jython(仅支持 Python2)。这些方式虽可快速上手,但难以使用 Python 的丰富库。PyImageJ 则提供纯 Python 的 API,用户可以用标准 Python3 在 Jupyter 等环境中编写脚本,轻松调用 ImageJ 的函数。例如,通过ij = imagej.init() 初始化后,可在 Python 中使用 ij.py.to_java(array) 将 NumPy 数组转换为 ImageJ 数据,或用ij.py.from_java(dataset)将 ImageJ 数据转为 NumPy 数组。这一点使得 Python 程序员能像调用本地函数一样调用 ImageJ 功能,且代码可使用 pandas、matplotlib、scikit-learn 等工具进行后续处理。
「自动化处理能力:」 ImageJ 原生支持宏批处理,可以通过“Process > Batch > Macro”对话框或脚本模板,将相同操作应用于一组图像。然而其自动化往往局限于 ImageJ 环境中,且在无显示环境下运行时,原始 ImageJ 的某些功能受限(例如 ROI 管理器依赖 GUI)。PyImageJ 则可在完全无 GUI 的环境中运行(默认模式即为无头 Headless 模式),能够在服务器或高性能集群中通过 Python 循环、函数或自动化框架(如 Snakemake、Airflow)批量处理图像。用户可以编写 Python 脚本循环读取、处理并保存图像,或结合 CellProfiler 等工具构建复杂管道。此外,PyImageJ 允许在 Python 脚本中直接使用 ImageJ 的命令和插件,例如调用图像拼接、滤波、分割等,一站式完成传统 ImageJ 和现代 Python 算法的混合处理。
「跨平台支持与部署:」 两者均具有优秀的跨平台能力。传统 ImageJ(和 Fiji)基于 Java,支持 Windows、macOS、Linux 等操作系统,甚至有面向 Raspberry Pi 和 Android 的版本。安装方式上,ImageJ 以可执行包或 ZIP 形式发布,可单独运行。PyImageJ 则作为 Python 包发布,可通过 conda 或 pip 安装,在上述平台上同样可用。更灵活的是,PyImageJ 支持在 Jupyter Notebook、Google Colab、Docker 等环境中动态初始化 ImageJ,方便与云计算和容器化部署集成。不过需注意,为了可重现分析结果,PyImageJ 文档建议固定 ImageJ 版本(例如 imagej.init('2.14.0'));而传统 ImageJ 通常依赖内置更新系统手动升级。
「与数据科学工具的集成:」 这是 PyImageJ 的显著优势之一。PyImageJ 设计用来打通 Python 科学计算生态与 ImageJ 插件生态。通过 imglyb 层,PyImageJ 在 Python 和 Java 之间提供了零拷贝的图像数据共享,实现了 ImageJ 图像与 NumPy 数组/xarray 数据的无缝转换。因此,用户可在 Python 中用 NumPy/Pandas 对图像数据进行分析统计,用 Matplotlib/Seaborn 可视化结果,用 scikit-image、ITK、OpenCV 等处理图像。例如,上述 Napari 示例中,PyImageJ 先用 SCIFIO 加载图像并转为 NumPy,随后直接交给 Napari 可视化。而传统 ImageJ 在这方面较弱:其宏或 Java 插件无法直接调用 Python 库,且不支持在 Jupyter 等现代平台直接使用第三方数据包。
「科研工作流程支持:」 PyImageJ 与现代科研流程高度契合。由于基于 Python,PyImageJ 的脚本和环境可轻松纳入版本控制与共享(如在 GitHub/Binder 上托管 Notebook 环境),并固定依赖包以保证可重现性。用户还可在 Notebook 中记录完整处理流程,实时可视化结果,并与他人协作。传统 ImageJ 虽有宏记录器和脚本编辑器,但宏语言不利于复杂控制结构,且宏脚本本身在不同环境中可能因 ImageJ 版本差异而表现不一。PyImageJ 既继承了 ImageJ 丰富的可视化能力(交互式多通道查看、3D可视化等),也能利用 Python 的显示手段进行图形输出。例如,PyImageJ 提供了将图像展示为 Matplotlib 图的接口(ij.py.show()),并可调用 Napari 等高性能查看器。综上,PyImageJ 更易于构建可复现、版本化管理的现代化分析流程。
科研群体与典型应用场景
PyImageJ 的设计初衷即服务于「算法开发者」和「实验科学家」双重需求。生命科学领域(如细胞生物学、神经科学和病理学等)长期依赖 ImageJ 进行显微图像分析,而近年来图像处理新方法多在 Python 中实现,因此这类领域的研究者往往倾向于使用 PyImageJ 来整合这两者。此外,图像算法开发者和数据科学家也会使用 PyImageJ:他们可以用 Python 调试新算法,同时调用 ImageJ 的成熟模块加速开发验证。生物医学工程和材料科学等领域的研究者,也可以利用 PyImageJ 结合现有的 ImageJ 插件完成特殊分析(例如组织配准、材料孔隙度分析等)。
「典型应用场景包括:」
- 「显微图像分割与计数:」 利用 PyImageJ 调用 ImageJ 的阈值、分水岭或 Trainable Weka 分割插件进行细胞/斑点检测(如官方示例中的 Blob Detection、Puncta Segmentation 等用例)。
- 「大规模图像拼接与配准:」 在组织学或高通量成像中使用 ImageJ 的 Grid/Collection Stitching 和 SIFT 配准插件,结合 Python 脚本处理批量切片(LOCI 项目即用 PyImageJ 运行了 ImageJ 的图像拼接和 SIFT 注册插件。
- 「多孔材料和生物结构分析:」 如 PoreSpy 项目通过 PyImageJ 调用 ImageJ 滤波器处理三维多孔材料影像;动物或植物组织中基于深度学习的结构化分析结合 PyImageJ 实现复杂预处理。
- 「病理切片超分辨重建:」 利用卷积神经网络对全片图像进行单幅超分辨(WSISR项目),结合 PyImageJ 和 Python 库完成色彩归一化和重建。
- 「交互式多维可视化:」 在 Jupyter+Napari 中通过 PyImageJ 读取多通道时序图像,实时探索和注释数据,实现数据科学工具(Napari、Pandas等)与 ImageJ 功能的结合。
以上示例表明,PyImageJ 在需要同时利用 ImageJ 现有算法和现代 Python 数据处理的场景中最具优势,尤其适合生命科学研究者、图像算法工程师和数据科学家等群体。
Follow && Sponsor
Sponsor
Follow ME
If you like us and use WeChat OR 微信, please follow our WeChat Official Account/微信公众号 - 「AllLink-official」 to get the latest updates.

评论
发表评论